多重成像与人工智能分析的整合应用
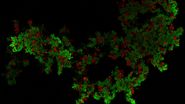
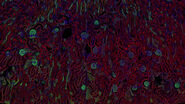
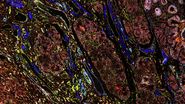
本研究探讨了癌症进展与治疗过程中免疫系统与肿瘤细胞之间复杂的相互作用关系。Cell DIVE 多重成像技术能在单张组织切片上检测数十种生物标志物,提供展示各类细胞类型的全组织分子图谱。Cell Signaling Technology 提供的 IHC 验证抗体与 Cell DIVE 完美兼容,可增强组织样本中各类细胞的检测与表型分析。Aivia 软件的整合通过 AI 引导的分割、表型分析和聚类分析简化了空间生物学工作流程。以 4T1 小鼠肿瘤模型为例的研究证实了该空间蛋白质组学工作流程的实用性,揭示了肿瘤内部异质性免疫景观及复杂的空间关系。
通过 Aivia 平台进行 AI 辅助分割与表型分析,我们对 Cell DIVE 多重全组织图像中所有标志物的表达特征进行了系统表征(图 1)。