介绍
序列断层成像(AT)是一种用于细胞和蛋白质结构分析的高分辨率三维图像重建方法(见图1)。它利用扫描电子显微镜(SEM)对超薄(>20nm)的有序阵列,即在硅基质载体上收集的树脂包埋生物标本连续切片,进行成像 [1]。采用序列断层成像,可实现对许多不同类型的组织和蛋白质进行定量和体积结构分析。由于比传统的共聚焦显微镜具有更好的横向和空间分辨率,因此它能够更好地帮助观察和理解细胞和蛋白质结构。此外,采用序列断层成像,可通过部分自动化的方式检查细胞和蛋白质结构,从而提高产量。
淋巴结是淋巴系统的一部分,一般呈肾形,分布在动物或人的全身。淋巴结由淋巴管连接,清除周围组织中的异物,帮助启动免疫反应。淋巴结是研究免疫细胞(淋巴细胞、树突状细胞和巨噬细胞)相互作用,以及癌细胞扩散(肿瘤转移)的理想对象 [2,3]。精确检查淋巴结对这类免疫和癌症研究具有重要意义。
已利用全自动连续切片方案通过序列断层成像对小鼠的淋巴结进行了研究。
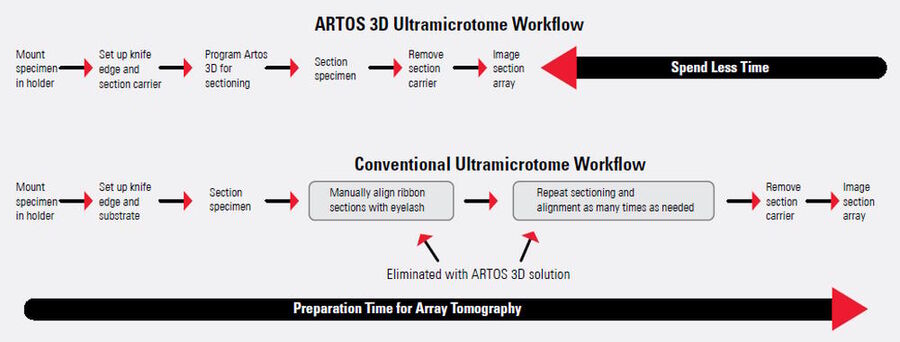
采用能够在排列整齐的阵列中实现均匀、超薄、高质量切片的全自动超薄切片机,连续切片提高了序列断层成像(三维图像数据采集)整个工作流程的效率(见图1)。徕卡显微系统公司的ARTOS 3D超薄切片机就是该连续切片方案的典型示例。该设备可自动产生数百个均匀的连续切片,并且由于采用了特殊设计的金刚石刀,避免了收集过程中切片带出现褶皱。另外,因在向基底载体上收集切片的过程中实现了较高的切片密度,从而节省了SEM设置的时间。此外,在利用玻璃基底制成的透明切片载体进行相关光电关联显微技术(CLEM)[4] 研究时,序列断层成像显示出其独特的优势,可以节省试样制备的时间。
材料和方法
淋巴结分离
取6-10周龄C57BL/6小鼠的腘窝淋巴结。按照Deerinck等人[5,6] 的OTO(锇-硫代碳酰肼-锇)和氰亚铁酸盐还原四氧化锇固定方案制备标本,提高扫描电镜成像时组织的对比度。
小鼠腹腔注射100mg/kg (wt/wt)克他命、10mg/kg (wt/wt)塞拉嗪和3mg/kg (wt/wt)乙酰丙嗪进行麻醉。然后经心灌注磷酸盐缓冲盐水(PBS) [0.025 M磷酸盐缓冲液,0.9% (wt/vol) 氯化钠, pH 7.4] 2分钟后,用含有2% (vol/vol)多聚甲醛和2.5% (vol/vol)戊二醛的0.1 M磷酸盐缓冲液(PB)溶液代替PBS灌注5-10分钟。灌注后立即收集淋巴结,并在室温(RT)下再固定2小时。
样本制备
固定和对比度增强
收集和固定后,在0.1M PB(pH 7.4)中清洗淋巴结标本。然后,为了在序列断层成像(AT)过程中增强对比度,将标本在含2% (wt/vol)四氧化锇(水溶液)和1.5% (wt/vol)亚铁氰化钾的0.1 M PB中在冰冻和避光环境中浸泡1小时。随后,用超纯水清洗(室温下5×3分钟),转到过滤过的硫代碳肼中,室温下水洗20分钟,然后再用超纯水清洗(室温下5×3分钟)。将标本置于2% (wt/vol)的四氧化锇(水溶液)中,室温下放置30分钟,然后再用超纯水清洗(室温下5×3分钟)。将其在1% (wt/vol)乙酸双氧铀(水溶液)中孵育,并置于4℃的冰箱中放置过夜。第二天,用超纯水清洗标本(室温下5×3分钟),在60℃的walton天门冬氨酸铅溶液中孵育30分钟,然后用超纯水清洗(室温下5×3分钟)。使用分级乙醇溶液(50%、70%、90%、96%和100%)进行脱水,每次5-10分钟,然后在无水丙酮中清洗(室温下2×10分钟)。
树脂包埋
将标本浸泡在溶解于丙酮(25%、50%和75%)的DurcupanTM ACM硬树脂中,分别浸泡1-2小时。然后,将其放在100%的Durcupan树脂中过夜,并转移到填充有新鲜树脂的BEEM胶囊中。DurcupanTM ACM树脂(Sigma-Aldrich)的4种成分(A-D)的配方为:A)11.4g, B)10g,C)0.3g,D)0.1g。最后,将树脂包埋的试样在60℃的烤箱中保存3天进行聚合。
标本剪切
用徕卡显微系统公司的EM TRIM2剪切装置对树脂包埋的小鼠淋巴结标本进行剪切(见图2A)。为在切片过程中形成整齐的条带,试样边缘必须保持平行。通常情况下,采用高速铣削系统,如EM TRIM2或EM Rapid方案,将试样进行剪切,使其边缘形成平行状。
试样切片
剪切完成的树脂包埋小鼠淋巴结试样采用徕卡显微系统公司的ARTOS 3D超薄切片机进行切片(见图2B)。使用ARTOS 3D方案可以实现高效切片工作流程的原因还包括:
- 刀台的电动运动和切片带间的自动转换(图3A)。
- 用于新样品的切片协议库
- 在准备大量的3D图像数据时,用于实际切片过程的试样自动剪切模式(图3B)
- 数字化横向测量功能;以及
- 选定刀口的自动靠近功能。
与传统的超薄切片机相比,ARTOS 3D方案实现的连续切片自动化大大提升了工作流程的效率。与传统的超薄切片机(如EM UC7系统和ARTOS 3D)在工作流程上的比较情况见图4。传统超薄切片机的工作步骤,ARTOS 3D方案中取消了这些步骤。
结果
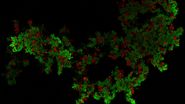
小鼠淋巴结研究的目标是探讨其皮质旁导管系统的复杂三维组织,该系统将过滤后的淋巴(含有小的可溶性抗原)从外周输送到静脉循环系统[10]。淋巴结的高分辨率3D图像重建最终应显示基质、骨髓和淋巴细胞如何在空间上相互作用并进入淋巴结导管系统。
采用阵列拓扑法重建了小鼠腘窝淋巴结旁的三维(3D)图像(见图5)。图像显示了在淋巴结细胞内迁移的T细胞的3D体积和形状及其亚细胞结构,如细胞核、线粒体和细胞质。
总结与结论
序列断层成像方法为高分辨率、可重复的3D图像重建开辟了道路,极大地推动了细胞生物学的发展。
采用ARTOS 3D超薄切片机,序列断层成像(AT)的试样制备和切片对齐变得更加简单,大大提升了整个工作流程的效率。效率的提升得益于其特别设计的金刚石刀、排水托盘和小艇,以便在切片载体上收集切片带而不产生褶皱。其薄片载体能够实现较高的切片带密度,并且尺寸较小,可以同时将多个载体以最佳方式装入SEM。因此,通过SEM可以对数百个切片进行成像,而无需重新加载。透明材料也可用于切片载体,因此ARTOS 3D超薄切片机也非常适用于相关光电关联显微技术(CLEM)[4]。
致谢
我们在此向奥地利科学技术研究所的Frank Assen、Ludek Lovicar、Vanessa Zheden和Michael Sixt表示感谢,感谢他们提供的序列断层成像图像数据。