使用AI准确检测多重图像中的细胞
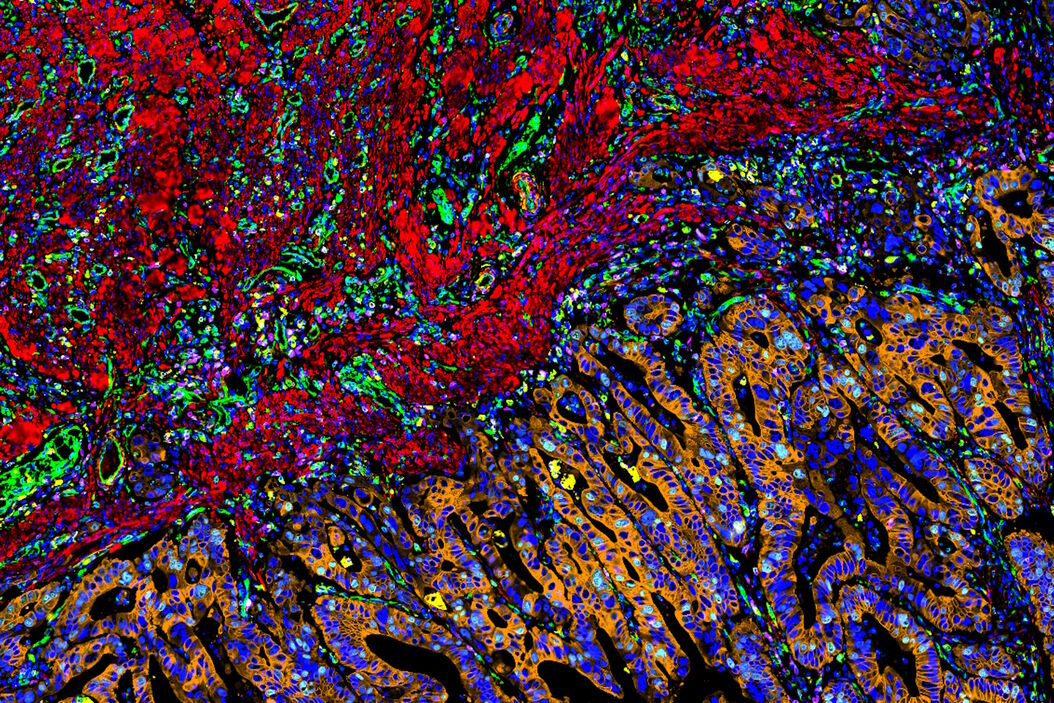
为了获得定量、生物学的见解,第一步通常是检测和分割整个图像中的单个细胞。Aivia的多重细胞检测配方利用了经过改进的Cellpose[1]算法,以准确检测具有不同形态学的细胞核和细胞膜。在此CAC(结肠腺癌)示例中,DAPI和NaKATPase通道分别用作细胞核和细胞膜标记物,以分割该组织中的所有细胞。在CAC组织中,总共识别并分割了555,480个细胞,平均细胞质面积为66.51 µm²,平均核面积为38 µm²,核圆度为0.77。
自动聚类揭示未知表型
在Aivia中,还可以使用自动聚类工具以无监督的方式识别未知的细胞表型。无需预先为每个表型定义生物标志物轮廓,PhenoGraph-Leiden [2] 聚类工具可以仅基于整个面板中生物标志物表达相似性来创建表型组。然后,这些表型集群可以根据它们与其他在分析中发现的集群的相似性,通过热图和树状图进行组织。利用树状图功能,我们可以直观地检查通常在异质CAC数据集中存在的一些重要趋势和富集的细胞群体。此外,这种分析还可以轻松地捕获组织中相对较少的细胞表型。